はじめに
こんにちは。サントリーの浜口です。デジタルテクノロジー分野で新卒入社し、現在データサイエンティストとして新たな知見を発掘しています。今回は、個人の健康に影響を与える腸内細菌叢を解析する中で、ぶつかった課題とその取り組みについてご紹介したいと思います。
背景と課題
現代社会において、健康への関心が高まる中、食品業界もヘルスケア分野への取り組みを強化しています。サントリーでも、飲料や食品の開発を通じて人々の健康に寄与することを目指し、その一環としてヘルスケアデータの解析を実施しています。特に、腸内細菌叢は健康状態や疾患リスクと密接な関係があることが明らかになっており、その解析は新商品の開発や健康増進の提案において重要な役割を果たします。
ヒトの腸内には、おおよそ1,000種類の腸内細菌が生息し、その組成は個人ごとに大きく異なります。 腸内細菌叢は消化や免疫機能、さらには精神状態にも影響を与えるとされ、解析することで個々の健康状態を深く理解する手がかりとなります。 腸内細菌データは非常に高次元で複雑です。そのため、直接全ての変数を解析するのは困難であり、膨大な次元の情報から本質的なパターンを抽出するためには、次元削減の手法が不可欠となります。 ここで、PCoA(Principal Coordinates Analysis、主座標分析)という手法が有用な解析手法となります。
腸内細菌叢解析におけるPCoAの重要性
PCoAは、多次元データを低次元の空間にマッピングすることで、サンプル間の類似性や違いを直感的に視覚化できる手法です。 たとえば、特定の食品を摂取したグループとそうでないグループの腸内細菌叢の違いをPCoAで表現することで、その食品が腸内環境に与える影響を容易に評価できます。
さらに、健康な人と疾患を持つ人の腸内細菌叢を比較する際にも、PCoAは有用です。 サンプル間の配置から、疾患に伴う細菌群の偏りや変動が明らかになり、これらの知見は新商品の開発や健康志向を訴求する際の科学的根拠として大きな役割を果たします。
以上の理由から、PCoAは腸内細菌解析において不可欠なツールであり、細菌群の多様性やその変動を深く理解するために非常に重要です。
今回このPCoAを用いるにあたって取り組んだ課題
腸内細菌叢解析の分野では、長年にわたってR言語がスタンダードとして使用されてきました。Rは統計解析や生物情報学に特化した豊富なパッケージを備え、研究者やデータサイエンティストに広く支持されています。特に、微生物データ解析では QIIME や phyloseq など強力なツールが充実しており、多くの解析パイプラインがRベースで構築されています。
一方、近年のデータサイエンスの発展や機械学習の普及に伴い、Pythonがデータ処理の分野で主流のひとつとなっています。 私たちのプロジェクトでも「前処理や機械学習でPythonを使うのに、腸内細菌叢解析のためだけにRを併用しなければならないのは煩雑」という声が上がり、 同じ環境内でPCoAを含む一連の解析を完結させたいというニーズがありました。
しかし、実際にPythonでPCoAを行うとなると、既存のライブラリでは実行環境やPythonのVersionの違い、次元圧縮手法の違いなどいくつか課題が浮上しました。 たとえば、Python用の微生物解析ライブラリとしてscikit-bioが存在しますが、 動作させるためのOSの依存関係が厳しいなど、環境依存・設計上の問題がいくつもありました。これらを総合的に判断した結果、 「必要最低限のライブラリだけで動作するPCoAを一からフルスクラッチで実装しよう」という結論に至りました。
このようにPythonで軽量実装することで、解析環境をPythonに統一し、環境構築の手間を削減しながら機械学習ライブラリなどとの連携もスムーズに行うことを目指しました。
今回の取り組みで実施したこと
1.PCoAについての原理からの理解と算出ロジックの理解
まずは PCoAの原理と算出ロジックをしっかり理解するところから始めました。 これまでの解析では、PCoAをRのライブラリ任せで使っていたため、 「サンプル間の類似性や違いを表す距離行列をどうやって作成するのか」「固有値問題をどのように解いて主座標を求めるのか」「そもそも入力データはどうやって得られたデータなのか」 などの根本的な部分を改めて学ぶ必要がありました。 その際、PCoAのマニュアルや公開されている文献を参照しながら、最近話題の生成AIにも助けてもらい、計算過程や入出力形式を把握するよう努めました。
2.RからPythonへのコンバート
次に、RからPythonへのコンバートを進めるステップでは、RとPythonという異なる言語間で計算処理を移植するうえでいくつか課題が生じました。 具体的には、R側で使用していた関数やパッケージがPythonには存在しない場合もあるため、Pythonで汎用的に使えるライブラリを探し、その計算ロジックをどこまで合わせられるかを試行錯誤しました。 また、計算結果が安定しないこともあり、アルゴリズムの細部や数値処理を見直すことでRの動作に近づけるよう取り組みました。
3.ユーザ配布用にテスト
最後に、ユーザ配布用のテストとして、社内外での利用を想定したパッケージの動作検証を行いました。 どのOSやPythonバージョンでも動作するかを確認するだけでなく、さまざまなデータサイズやファイルフォーマットにも対応できるようにテストを重ねています。 実際にユーザの環境で起こったエラーの対処法をいくつも検討しながら、より汎用性が高い設計を目指しました。
結果の検証
実装したPCoAの結果の妥当性を確認するため、同じデータセットと距離指標を用いてRでのPCoA解析と比較しました。
検証には腸内細菌解析を支援するプラットフォームMANTAのデモデータ20行分をもとに合成した1000行分のサンプルデータを用いています。
今回検証用にRでのPCoA解析はveganパッケージを使用しています。距離行列はBray Curtis距離行列を用いています。
# RでのPCoA解析例
library(vegan)
data <- read.csv("Sample.csv", header=FALSE)
dist_matrix <- vegdist(data, method="bray")
pcoa_result <- cmdscale(dist_matrix, eig=TRUE, k=2)
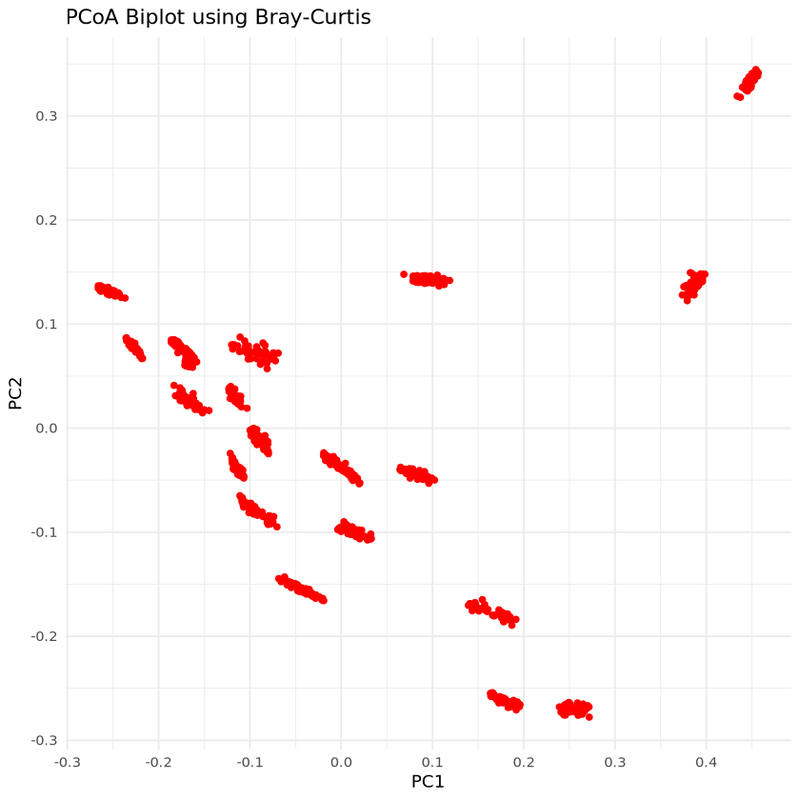
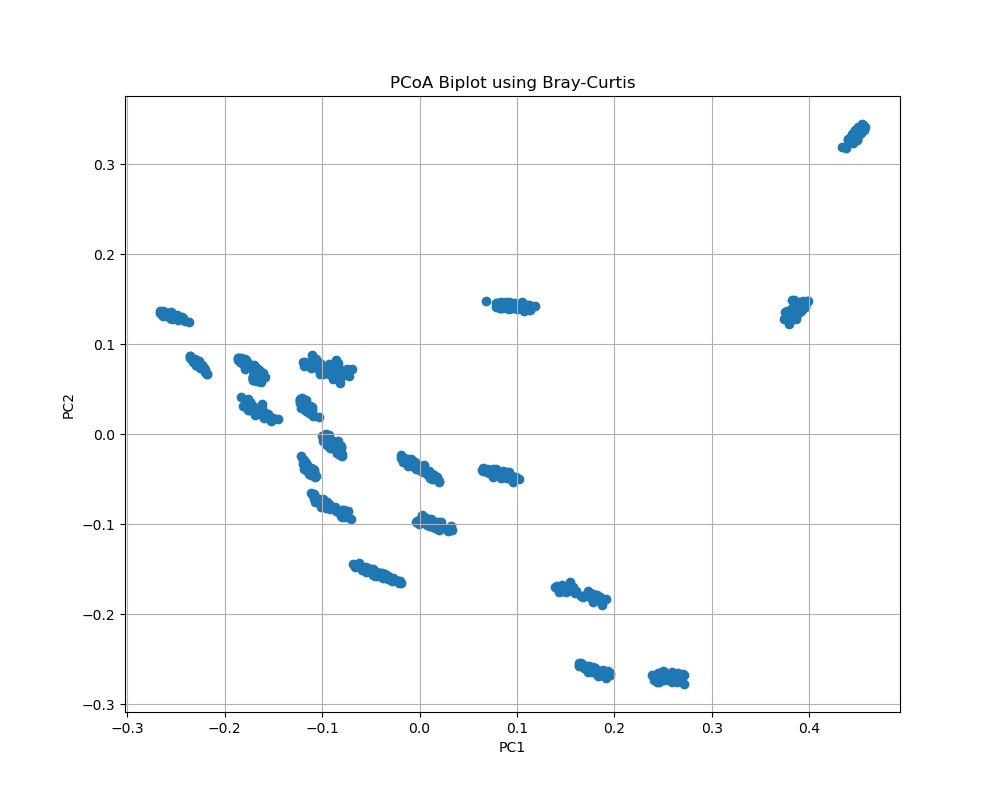
左がRの散布図、右の散布図結果です。
 |  |
|---|
RとPythonで得られたPCoAの結果を比較したところ、散布図の結果が一致していることを確認できました。
おわりに
サントリーでは、デジタル&テクノロジーの分野で共に成長する仲間を募集しています。 私たちは、最新の技術を駆使して新しい価値を創造し、社会に貢献することを目指しています。 また、ヘルスケアデータ解析やバイオインフォマティクスの分野においても、先進的な研究と実践を推進し、健康の未来を切り拓く取り組みを積極的に行っています。 興味をお持ちの方は、採用サイトおよび新卒採用ページをご覧ください。
なお、今回作成したパッケージについては次の記事にて詳細を解説いたします!